非因解读| Digital Spatial Profiler数字空间多组学技术的应用
Digital Spatial Profiler
数字空间多组学技术的应用
非因生物的数字空间多组学分析系统(Digital Spatial Profiler,DSP)是针对肿瘤免疫和肿瘤微环境高精度、多维度分析的新一代空间组学技术,可以在单张切片上通过形态学指导选择兴趣点(Region of Interest,ROI),来实现基于每个ROI微环境的90重蛋白或>1,800重癌症转录组的原位表达谱分析,而且很快将实现18,000重全转录组的原位分析,其定量效果、通量和可靠性远超传统的以荧光信号为基础的多重免疫组化与多重免疫荧光技术。
随着癌症免疫治疗的蓬勃发展,越来越多的研究发现深入了解细胞亚群的空间分布以及特定蛋白和RNA的空间表达,可以为深入研究肿瘤微环境并阐明生物学机理提供深层次的信息,也为基于微环境的生物标志物开发带来了新的研究方向。目前新兴的空间转录组学技术方法大部分还停留在实验室阶段(FISSEQ,MERFISH,seqFISH,Slide-seq,HDST,GEO-seq等),不适用于商业化。而目前商业化的技术大部分只适用于冷冻新鲜组织的RNA,受限于石蜡切片,且无法做到深入了解肿瘤微环境的相关组织信息来对研究策略进行预判。
非因生物基于空间多组学DSP技术搭建了一套完整的空间多组学方案,解决了肿瘤微环境研究的难点与痛点。DSP技术的基本流程如下:
1.寡核苷酸通过可紫外线光裂解接头共价偶联一抗(如图1a)或mRNA杂交探针进行原位捕获;
2.通过形态学指导进行ROI(兴趣区)划分后,对ROI进行紫外光解并收集兴趣点区域靶标上的寡核苷酸序列;
3.解离的寡核苷酸再通过杂交法经由nCounter计数或采用建库方法实现与二代测序的接头,从而对蛋白质或RNA进行高通量检测(图1a)。
非因生物DSP空间组技术从样本上机到数据输出的整个工作流程(图1a)可以在2.5天内完成,并衔接后续成熟的生物信息学流程可在一周内完成分析报告,简单高效。


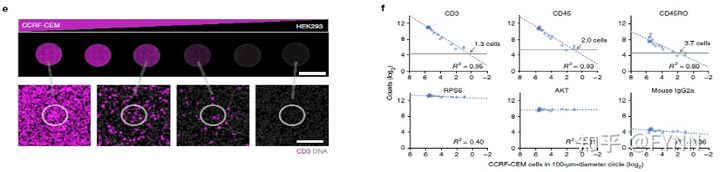
图1
今年5月份由Beechem博士联手Mills团队对DSP技术进行了蛋白与转录组在FFPE组织中的全方位测试,本次就是对其发表在Nature Biotechnology(IF:36.558)上的“Multiplex digital spatial profiling of proteins and RNA in fixed tissue”文章进行解读(图2)。

图2
团队用IHC比较扁桃体上CD3和B7-H3的未偶联寡核苷酸抗体和偶联寡核苷酸抗体的染色特异性,发现染色结果高度相似(图1b)。选择稳定表达的细胞系制作TMA芯片,用CD3、CD45和PanCK荧光抗体和核染进行原位标记,检测了41个蛋白靶点的原位表达情况,得到23个形态学划分的ROI区域(图1c)。通过对所测细胞系中的6个蛋白靶点表达量的分析,发现DSP计数具有广泛的信号动态范围,阴性和阳性细胞株有不同水平的表达差异(图1d)。接下来将CD3阳性CCRF-CEM细胞按比例加入CD3阴性的HEK293细胞制作TMA切片,选取100微米直径的ROIs对细胞计数,发现随着CCRF-CEM细胞比例下降,CD3荧光强度亦呈相同比例下降,同样的结论也在CD45、CD45RO两个靶标中得到重现(图1f)。该实验验证了DSP可以根据已知的细胞数量,通过ROI来量化蛋白质的相对水平。
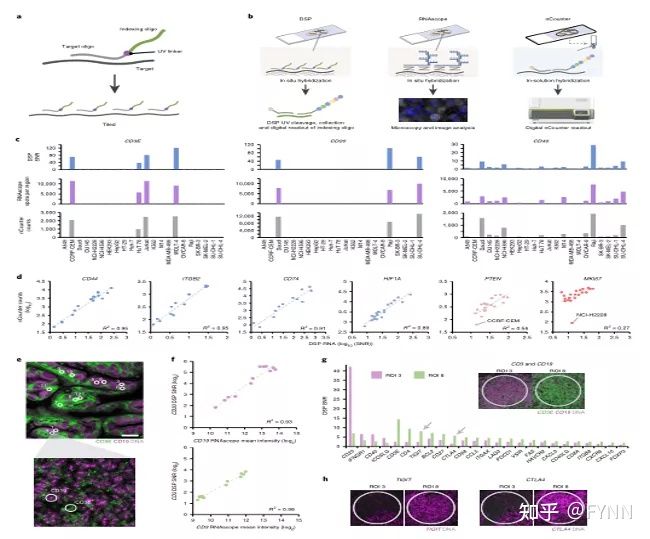
图3
在空间转录组方面,团队检测所用的寡核苷酸探针是由三部分构成,包括与检测靶点对应的一段互补序列、可光解连接子和一段特定顺序的寡核苷酸序列。将22种细胞制成TMA切片,对三种检测方法进行对比:从左向右依次是DSP、RNAscope、nCounter(图3b)。发现三种方法在CD3E、CD20和CD40三个靶标定量高度一致(图3c)。将DSP和nCounter进行比较,发现具有较好的相关性(图3d)。在石蜡样本中,研究者对DSP和RNAscope FISH荧光标记的CD3E及CD19进行比较(图3e),发现具有较高的一致性(图3f)。接下来选择了两个代表性的区域—T细胞富集区及B细胞富集区,如预期B细胞富集区可看到CD20以及在B细胞中特异表达的其他靶标的高计数,T细胞富集区域也得到了相似的结论(图3g)。RNAscope对这两个代表区进行验证结果显示,在CD3E高表达的区域中TIGIT和CTLA4转录本富集,该结论与DSP的数据结果具有高度一致性。
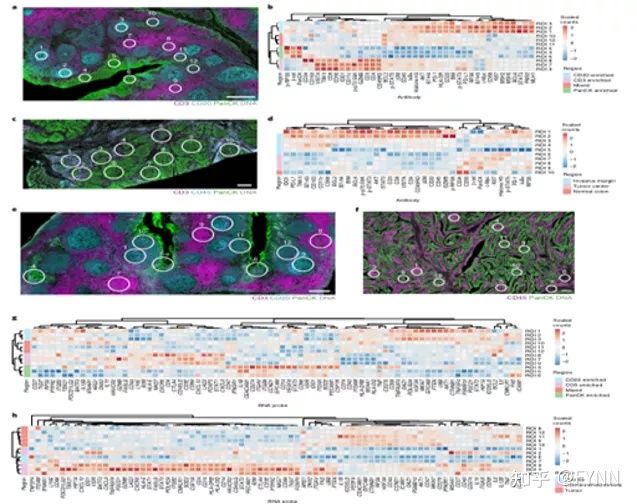
图4
接下来,研究者使用CD3(红色)、CD20(青色)、PanCK(绿色)和DNA(灰色)对扁桃腺切片进行四色荧光成像 (图4a)。选择了涵盖一系列形态组织特征的12个ROI进行44重蛋白靶标分析,发现通过形态学抗体标记富集区域DSP对应靶标计数呈正相关 (图4b)。采用同样策略,同时分析结肠直肠癌组织(图4c),选择肿瘤中心、浸润边缘及正常组织区域进行聚类分析。如预期,浸润边缘的免疫靶标表达水平最高,而肿瘤中心则与肿瘤标志物相关,如PanCK、β2M和Ki-67(图4d)。作者还对原位RNA表达进行了类似的分析,同样得到了相似的结论(图4e–h)。
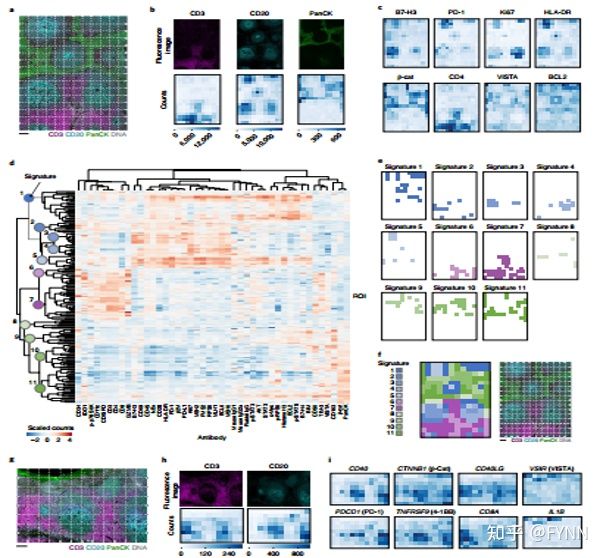
图5
接下来,作者团队以网格方式划分组织,选择彼此相邻的方形ROI,得到一个集中区域(图5a)。发现每种形态学标记的分布可以通过DSP实现组织原位表达重现(图5b)。41种抗体在可视的形态学标记下可以显示出不同的蛋白空间表达模式(图5c)。为了对这些网格数据进行无偏倚分析,对所有ROI和靶标进行了分层聚类热图分析(图5d)。选择特定靶标并作为唯一的标记显示,每个靶标用与DSP分析相同的网格格式显示(图5e)并进行组合,发现组成的图像与采用荧光靶标呈现的组织图像相似。同样在RNA分析中观察到相似的结果,DSP重现了扁桃体免疫生物学的表达谱空间分布情况(图5g-i)。
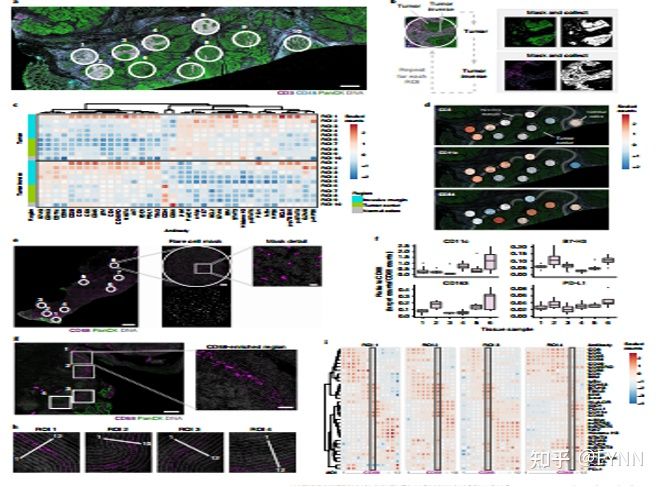
图6
为了进一步验证DSP基于组织结构获得生物信息的能力,研究者将图4c的每个ROI划分为肿瘤细胞和肿瘤微环境(TME)两个部分(图6a)。将同一ROI内的PanCK阳性、阴性区域采用Masking覆盖方式分别收集靶标(图6b),发现两种区域显示不同的表达谱,免疫相关靶标在肿瘤微环境或基质细胞中强烈富集,高浸润肿瘤区域免疫标记物含量最高(图6c),将靶点信息对应到组织图像ROI(图6d),可看到免疫标志物的富集与组织区域信息具有相关性。团队继续对IBD(一种肠炎症疾病)的特殊细胞及免疫细胞区域进行区域划分,并对巨噬细胞富集区域(CD68荧光指导)进行高维度分析(图6e-i),得到不同组织区域的CD68及其他靶标计数信息(图6e,f), 发现CD68高表达与肠上皮组织轮廓具有相关性(图6g),以此划分研究区域(图6h)。利用这种方法,研究者可以在CD68 (巨噬细胞)富集的基础上识别出三个不同的免疫生物学组织亚区域,在IBD样本的上皮细胞中找到具有明确的效应物和相关的细胞因子信号,这可能是造成炎症反应失调的一部分原因。
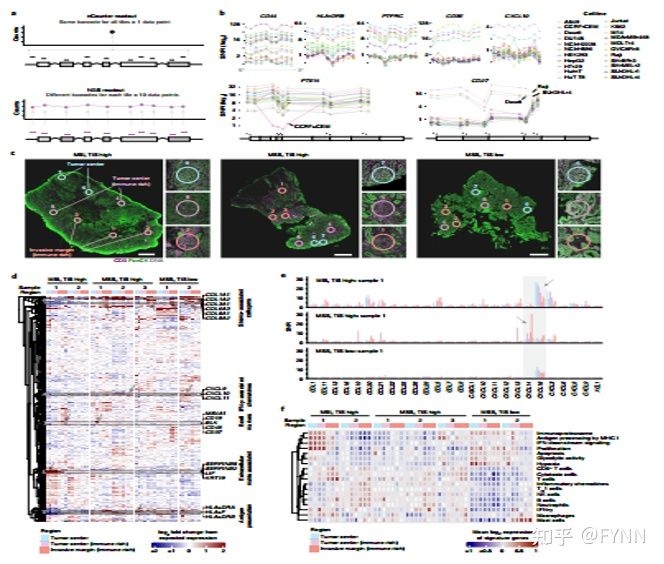
图7
为了增加DSP靶标检测上限,同样可以使用二代测序(NGS)作为下游定量方式。与nCounter不同的是,NGS可对应到每个RNA转录本的每个靶标片段(图6a,b),而且两种定量方法具有高度的一致性。通过研究1412个(4998个探针)基因原位表达在FFPE切片组织上,研究者分别选择了具有不同微卫星稳定状态(MSS或MSI)或肿瘤炎症(TIS)三种不同组织表征 (图6c) ,分析了肿瘤和免疫细胞丰度(图6d)。同时对趋化因子进行分析,发现在MSS组织CXCL14富集和在MSI组织CXCL16富集(图6e),同时根据生物过程特征基因的差异表达刻画了样本与生物过程相关程度的聚类热图(图6f)。研究证明了在FFPE样本上针对微环境进行复杂通路的空间基因表达分析以及对关键信号分子(如趋化因子)进行靶向分析的可行性。
总结
非因生物的空间多组学DSP技术不仅适用于冰冻样本,同时广泛适用于FFPE样本的蛋白质或RNA空间微环境分析。该技术能实现目标区域的单细胞群的分析及其靶向捕获的方式,而且其RNA检测灵敏度可低至约600个mRNA转录子,不需要很高的测序深度也可实现非偏倚捕获低丰度转录本。目前该技术已经实现18,000重全转录组分析(Whole Transcriptome Atlas, WTA),针对人类和小鼠全转录组的分析,将在2021年第一季度全面推向科研用户。
本文来自互联网用户投稿,文章观点仅代表作者本人,不代表本站立场,不承担相关法律责任。如若转载,请注明出处。 如若内容造成侵权/违法违规/事实不符,请点击【内容举报】进行投诉反馈!