SRA原始数据下载-aspera-ascp命令Mortix
一、前置环境
win11子系统Ubuntu 20.0
安装aspera-cli (aspera的升级版)
conda install -c hcc aspera-cli
安装后需要找到asperaweb_id_dsa.openssh
/home/yang/miniconda3/pkgs/aspera-cli-3.9.6-h5e1937b_0/etc/asperaweb_id_dsa.openssh
二、数据下载
2.1 利用ascp下载
下载SRR_list
NCBI-All Databases 输入PRJN或SRP或SRR号
如人SRP048601
点击SRA
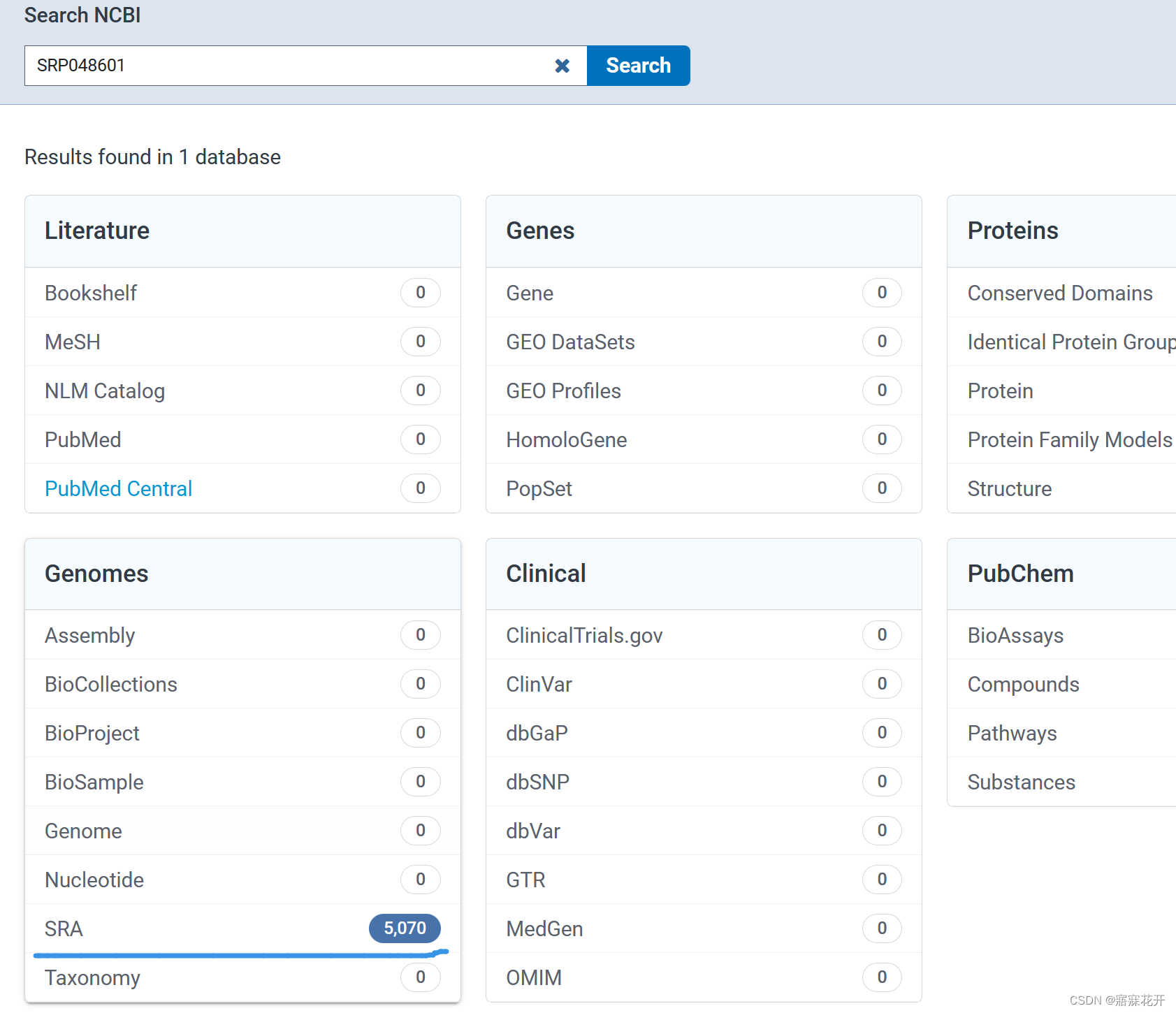
点击 Send results to Run selector
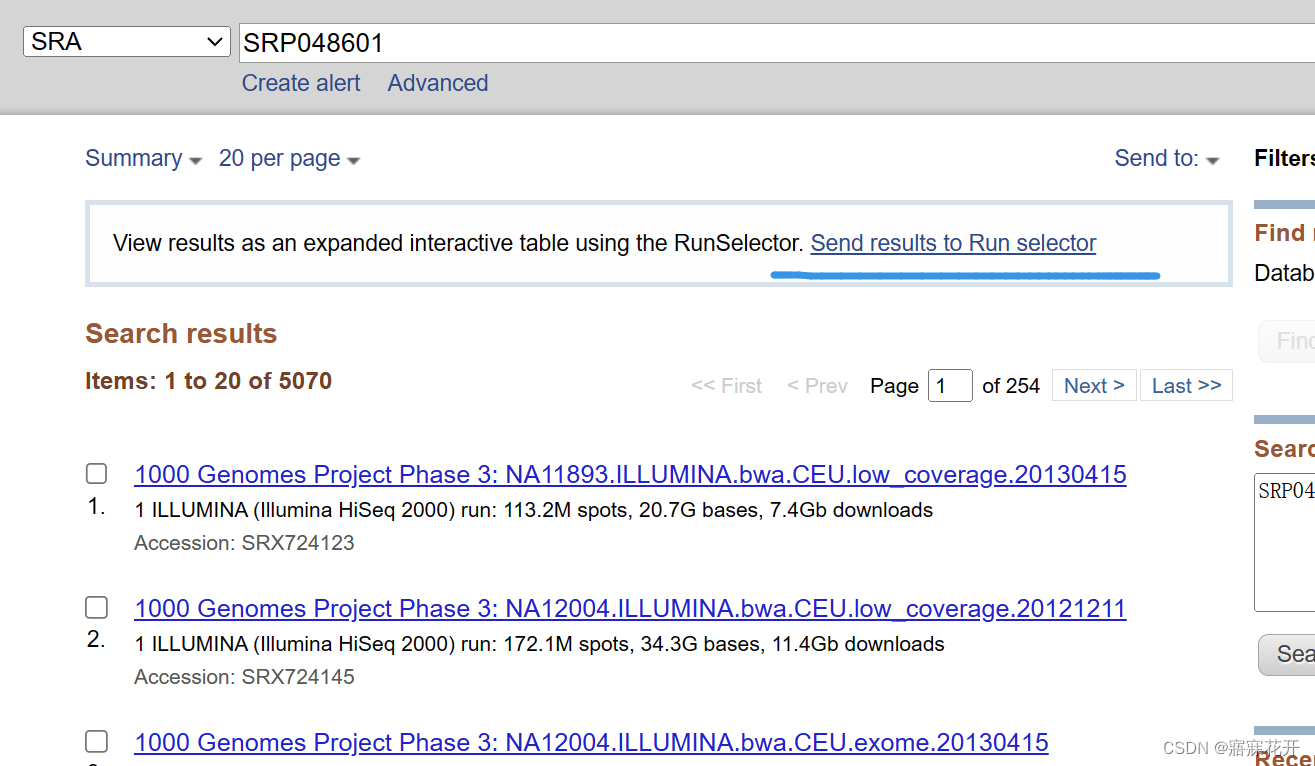
勾选需要的个体或全选,然后点击Accession List,其中Metadata下载的是完整信息列表
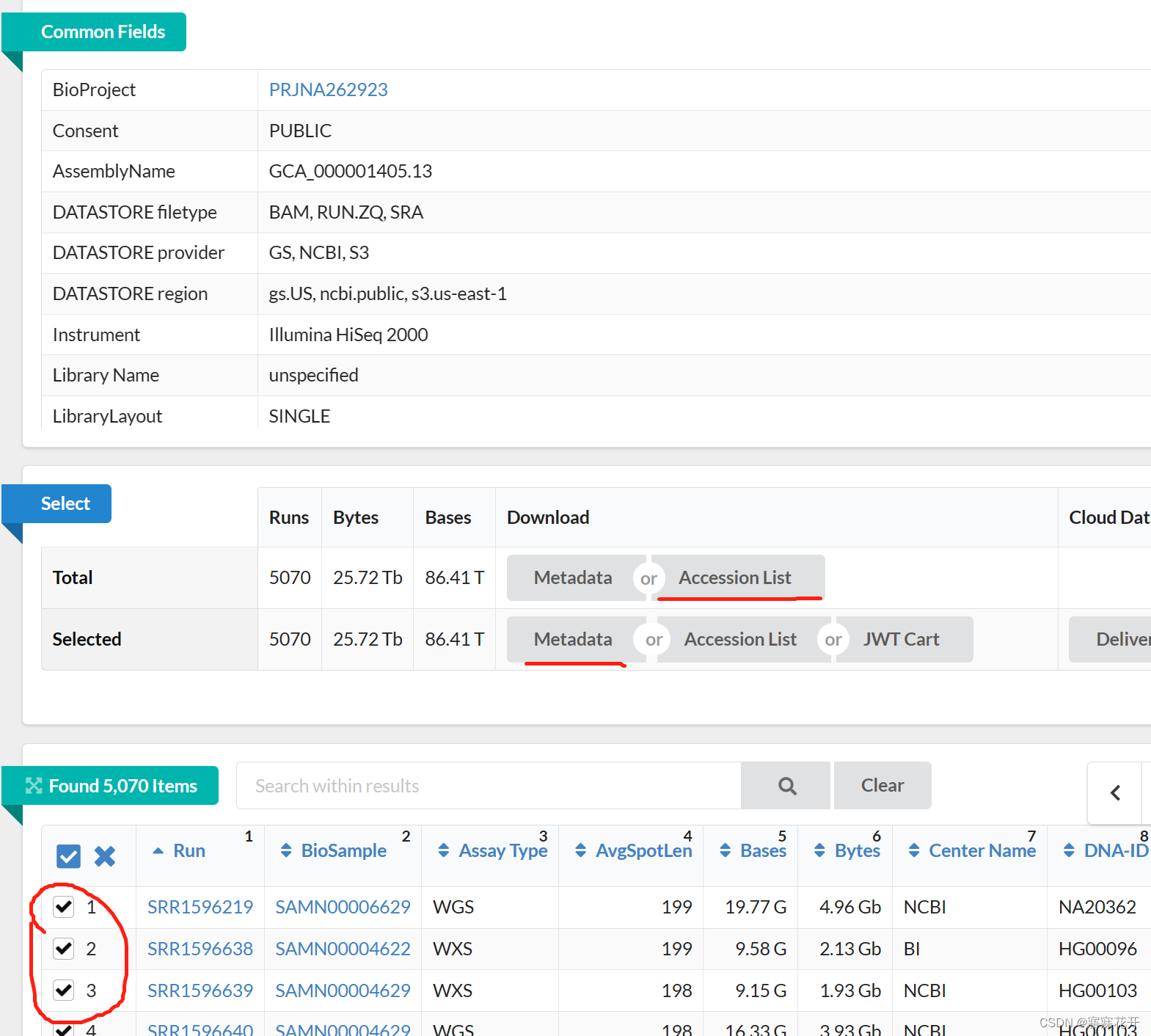
SRR+6位、7位或8位数均可用以下代码
ascp下载代码
echo SRA_datadown
openssh=/home/yang/asperaweb_id_dsa.openssh
cat SRR_list |while read id
donum=`echo $id | wc -m `if [ $num -eq 12 ]then dateecho "SRR + 8"x=$(echo $id | cut -b 1-6)y=$(echo $id | cut -b 10-11)echo "Downloading $id "(ascp -QT -l 300m -P 33001 -k 1 -i $openssh era-fasp@fasp.sra.ebi.ac.uk:vol1/fastq/$x/0$y/$id/ ./)elif [ $num -eq 11 ]then dateecho "SRR + 7"x=$(echo $id | cut -b 1-6)y=$(echo $id | cut -b 10-10)echo "Downloading $id "( ascp -QT -l 500m -P33001 -k 1 -i $openssh era-fasp@fasp.sra.ebi.ac.uk:vol1/fastq/$x/00$y/$id/ ./)elif [ $num -eq 10 ]thendateecho "SRR + 6"x=$(echo $id |cut -b 1-6)echo "Downloading $id "( ascp -QT -l 500m -P33001 -k 1 -i $openssh era-fasp@fasp.sra.ebi.ac.uk:vol1/fastq/$x/$id/ ./ )fi
done
注意:SRR_list行尾应有$
具体可用cat -E file查看
有^M的可通过dos2unix file 去除
此外,由于ascp部分数据下载不全,需设定定时重启该脚本以确定每一个数据的完整性(断点续传
-k 1)
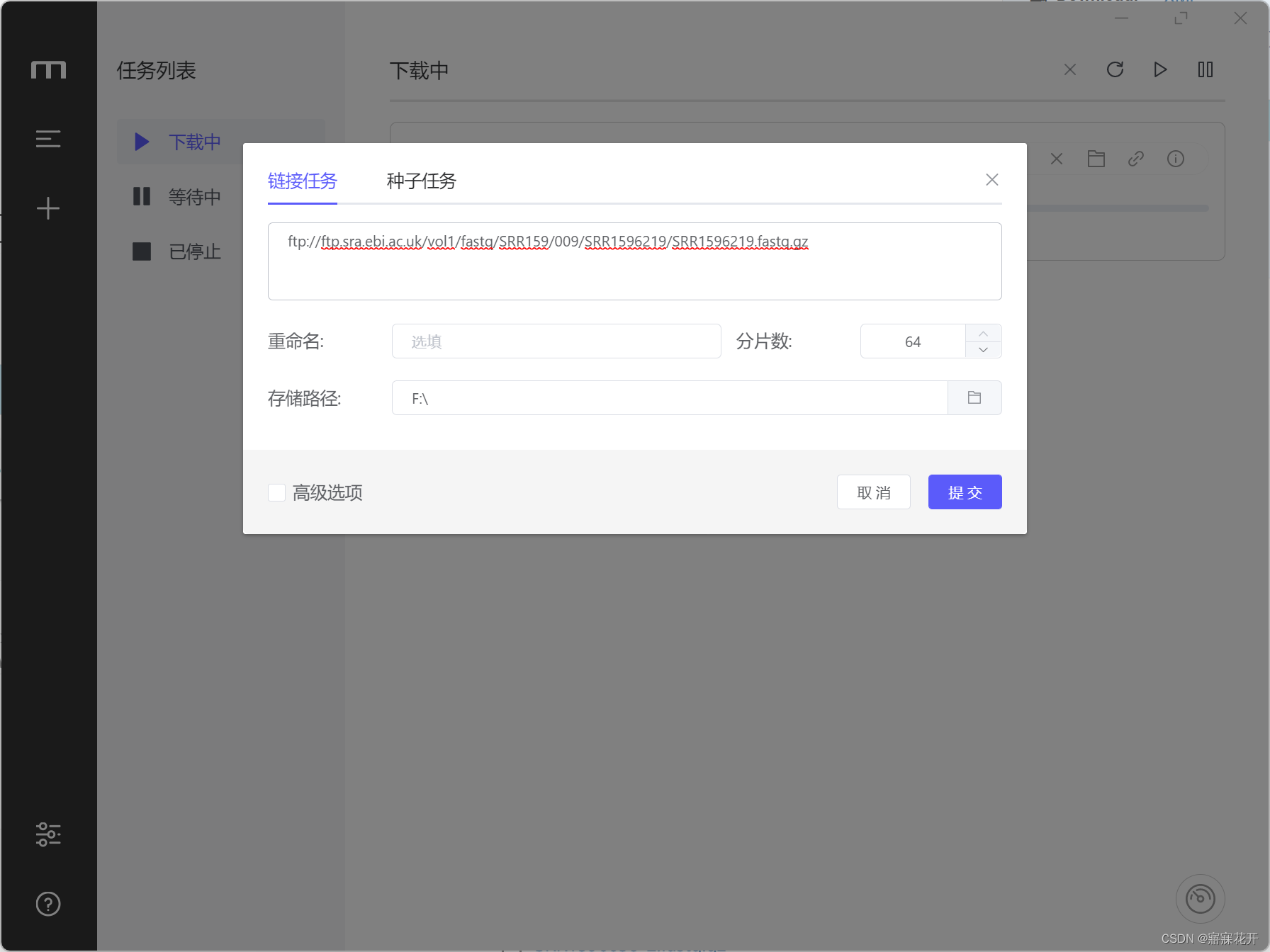
2.2 Mortix
ascp有时因服务器维护不可用,直接使用Mortix下载ftp,容易中断需手动恢复,或者使用Linux的wget 断点下载
先在ENA 中搜索PRJN或SRP或SRR
右击获取连接
或者直接利用代码批量生成ftp链接
echo SRA_datadown
openssh=/home/yang/asperaweb_id_dsa.openssh
cat SRR_list |while read id
donum=`echo $id | wc -m `if [ $num -eq 12 ]then dateecho "SRR + 8"x=$(echo $id | cut -b 1-6)y=$(echo $id | cut -b 10-11)echo "Downloading $id "echo ftp://ftp.sra.ebi.ac.uk/vol1/fastq/$x/0$y/$id/{id}_1.fastq.gzecho ftp://ftp.sra.ebi.ac.uk/vol1/fastq/$x/0$y/$id/{id}_2.fastq.gzelif [ $num -eq 11 ]then dateecho "SRR + 7"x=$(echo $id | cut -b 1-6)y=$(echo $id | cut -b 10-10)echo "Downloading $id "echo ftp://ftp.sra.ebi.ac.uk/vol1/fastq/$x/00$y/$id/{id}_1.fastq.gzecho ftp://ftp.sra.ebi.ac.uk/vol1/fastq/$x/00$y/$id/{id}_2.fastq.gzelif [ $num -eq 10 ]thendateecho "SRR + 6"x=$(echo $id |cut -b 1-6)echo "Downloading $id "echo ftp://ftp.sra.ebi.ac.uk/vol1/fastq/$x/$id/{id}_1.fastq.gzecho ftp://ftp.sra.ebi.ac.uk/vol1/fastq/$x/$id/{id}_2.fastq.gzfi
done
参考
Aspera——利用SRR号批量高效下载FASTQ或SRA数据 - 知乎 (zhihu.com)
在Linux中如何去除文档里^M?_E.die的博客-CSDN博客_文档去除m
本文来自互联网用户投稿,文章观点仅代表作者本人,不代表本站立场,不承担相关法律责任。如若转载,请注明出处。 如若内容造成侵权/违法违规/事实不符,请点击【内容举报】进行投诉反馈!